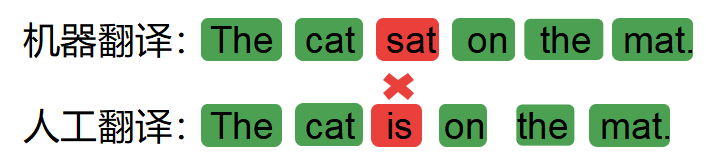
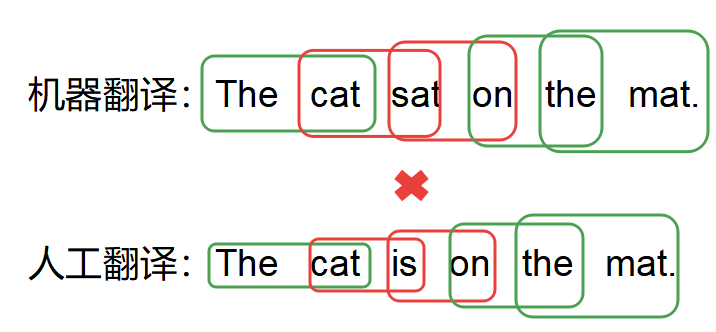
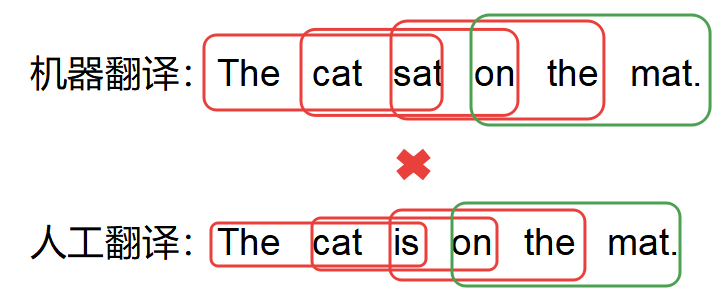
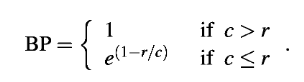最近有一个医疗图像的比赛,医疗图像以前没有接触过,对于这种格式的数据不甚了解,做了一些调研,记录一下。
定义
医疗数位影像传输协定(DICOM,Digital Imaging and Communications in Medicine)是一组通用的标准协定,在对于医学影像的处理、储存、打印、传输上。它包含了档案格式的定义及网络通信协定。DICOM是以TCP/IP为基础的应用协定,并以TCP/IP联系各个系统。两个能接受DICOM格式的医疗仪器间,可借由DICOM格式的档案,来接收与交换影像及病人资料。—— 维基百科
简言之,DICOM就是一种医疗行业使用的一种数据存储传输标准,广泛应用于各种医疗设备,如:CT、核磁共振、超声等。
内容
协议包含编码格式,图像格式、仪器厂商、仪器序列号、数据生成时间、数据采集方式、病人性别、病人年龄等等。下面是使用PYDICOM读取数据打印的结果。
1 | (0008, 0005) Specific Character Set CS: 'ISO_IR 100' |
具体内容并不关心,只看看那些可能会用到,比如病人性别、年龄。
读取显示
使用pydicom
1 | #-*-coding:utf-8-*- |